质谱指纹图谱专家共识 | 蛋白/多肽指纹图谱在肿瘤中的临床应用专家共识(2024版)
摘要:随着精准医学的发展,蛋白/多肽指纹图谱在肿瘤诊断、治疗选择和预后评估中的价值日渐突出。本专家共识旨在全面梳理蛋白/多肽指纹图谱的检测分析方法和在肿瘤临床领域中应用的潜力,围绕蛋白/多肽指纹图谱的定义、临床意义、检测技术、数据分析方法,以及在肿瘤诊断、治疗决策和复发监测中的应用,结合最新研究进展,提出一系列关于蛋白/多肽指纹图谱检测和应用的共识推荐,旨在提升医疗企业从业人员和临床医技人员对其的认知水平,促进蛋白/多肽指纹图谱相关检测产品的研发、转化和在临床肿瘤诊疗全流程中的应用,从而为患者提供更为精准的治疗方案。
在现代肿瘤精准检测领域,蛋白/多肽指纹图谱在肿瘤的诊断、治疗决策和预后评估中扮演着越来越关键的角色。为了推动蛋白/多肽指纹图谱在肿瘤临床管理中的应用,提高医疗企业从业人员和临床医技人员对其重要性的认识,同时为蛋白/多肽指纹图谱相关检测产品的研发、转化和临床应用提供指导性建议,确保检测结果的准确度和可靠性,基于最新的研究成果,本领域内的专家学者共同编写了本专家共识,系统性地介绍了蛋白/多肽指纹图谱的基本概念、检测分析技术及其在肿瘤诊断、治疗决策、疗效和预后评估中的作用,以促进该领域的标准化发展,最终实现个性化诊疗策略的优化,为患者的精准诊疗提供决策依据。根据相关领域的研究进展,本专家共识将及时进行修订,以满足肿瘤临床应用的需要。
1. 蛋白/多肽指纹图谱概述
多肽通常指由2~100个氨基酸组成的小分子蛋白(相对分子质量200~10 000)[1],主要由基因表达和蛋白代谢产生,如细胞因子、生长因子、激素和神经肽等,包含基因表达信息、结构蛋白和分泌蛋白信息及酶谱水平和活性信息等,处于中心法则下游,与表型关系更加密切[2]。目前研究显示,多肽广泛参与生物体内的生理和病理过程。在肿瘤的发生和发展过程中,多肽也发挥关键作用,通过调控转录、转录后、蛋白-蛋白互作、酶活性、信号传递和细胞间通讯等多种机制影响肿瘤进展[3]。基于多肽包含丰富的生物学信息、具有组织与疾病特异性和在肿瘤发生与发展全流程中的重要作用,肿瘤相关多肽引起研究者的广泛兴趣[1,4]。特异性的肿瘤相关多肽可在肿瘤组织高压和血管高通透性的作用下由产生的部位快速穿过屏障进入体液中,用于肿瘤检测[1,5]。然而许多单一多肽或者蛋白质标志物用于疾病检测的准确度有限,尤其是面对分子机制复杂和异质性极强的肿瘤时,其应用受到限制。多肽组学是研究生物体内多肽组成、结构和功能的学科[1]。通过临床大队列的多肽组学研究可以筛选疾病相关差异多肽,进一步利用生物信息学方法整合多个疾病相关多肽或蛋白信息,形成蛋白/多肽指纹图谱模型用于疾病的检测和预测,可显著提高检测结果的准确度和检测人群的适用范围[6-7],因此可通过收集体液样本进行蛋白/多肽指纹图谱检测分析,用于肿瘤精准检测。相较于目前常见的其他液体活检技术,如循环肿瘤DNA(circulating tumor DNA,ctDNA)、循环肿瘤细胞(circulating tumor cell,CTC)、外泌体和微小RNA(microRNA,miRNA),蛋白/多肽指纹图谱在敏感度、早期检测准确度和应对肿瘤异质性等方面具有一定优势(表1)。指导意见1:多肽包含丰富的疾病相关信息,可用于肿瘤检测。基于多肽组学数据筛选,整合多个肿瘤相关多肽的蛋白/多肽指纹图谱较单一多肽,可为肿瘤检测提供更精准的结果。
2. 蛋白/多肽指纹图谱检测分析技术
2.1 临床样本的选择通常用于蛋白/多肽指纹图谱检测的样本包括体液、组织、原代细胞和培养液等,各种样本均有优势和不足。体液样本相对易获取,利于连续取样,同时一些体液样本具有自我平衡属性,检测结果的重复性大幅提高。而组织和原代细胞样本来源于特定组织区域,其蛋白/多肽指纹图谱和组织本身关系更强。然而在肿瘤管理过程中,内镜学、影像学和组织病理学等临床参考标准检测技术在准确度上具有明显优势,对于发挥辅助角色的分子检测技术,重点关注受检者依从性和样本易获取性等。因此本专家共识重点阐述各体液样本在蛋白/多肽指纹图谱检测分析中的特色。目前蛋白/多肽指纹图谱检测分析常用的体液包括血液、尿液、脑脊液和唾液等(表2)[8-15]。
血液样本主要包括血浆和血清,两者间多肽组存在差异[16]。严重溶血、严重脂血或严重黄疸样本均建议重新采集。获取血浆样本的采血管中抗凝剂种类包括肝素、乙二胺四乙酸(ethylene diamine tetra acetic acid,EDTA)和柠檬酸钠等,其中EDTA最常用,因为其可增强多肽稳定性[17]。采用真空血清采血管采集血液样本后,至血清分离的时间间隔不宜>6 h。如果条件限制,需要延迟分离,则建议采样后全血样本2~8℃保存,12 h内完成血清分离。血浆和血清转移时应尽可能避免吸入细胞或细胞碎片,分离前不可冷冻全血样本,以免溶血。血浆或血清分离后应在低温下保存和转运,如果不能立即开展后续处理,可将样本置于≤-70℃,期间避免反复冻融[18],以免对多肽组检测造成不利影响。采用血液样本的优势在于采样微创,各临床机构和健康管理中心经验丰富,样本可及性高。且血液样本中蛋白/多肽丰度较高,达到60~80 mg/mL,其中包含很多来自疾病组织分泌和转运的蛋白/多肽,适用于多数疾病检测,临床转化前景好[5]。然而血液样本中蛋白/多肽浓度变化范围较宽,>10个数量级[5]。这对多肽分离富集和检测技术提出了严峻挑战。
表1 蛋白/多肽指纹图谱和其他液体活检技术用于肿瘤检测的优势和不足
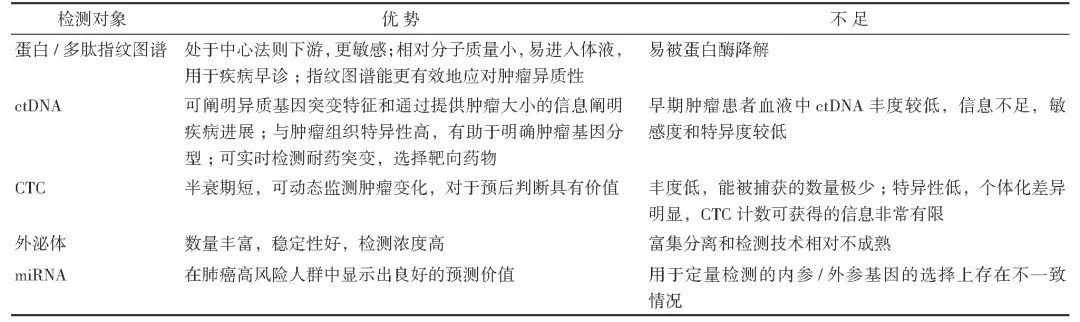
注ctDNA:循环肿瘤DNA;CTC:循环肿瘤细胞;miRNA:微小RNA(microRNA)
表2 蛋白/多肽指纹图谱检测常见体液样本的特征分析
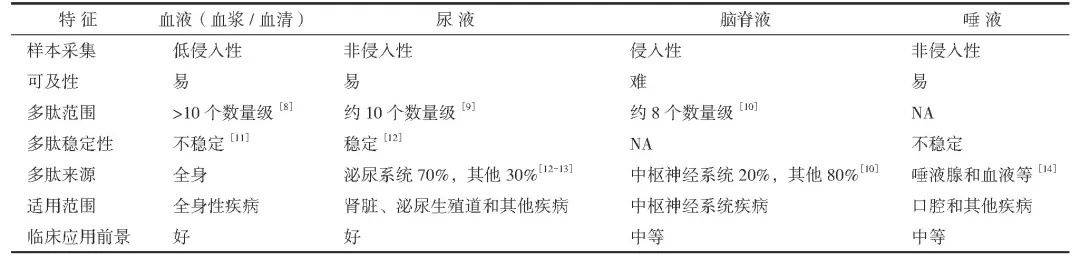
注NA:缺失值
尿液由肾脏生成,是经输尿管和膀胱排出的含有大量代谢终产物的液体。尿液中多肽约70%来自泌尿系统,尤其是肾和膀胱,因此可基于尿液蛋白/多肽指纹图谱检测泌尿系统疾病[19-20]。另外约30%的尿液多肽来自血液肾小球滤过,因此尿液蛋白/多肽指纹图谱同样可用于其他疾病的检测[21-22]。通常尿液在排出前已在膀胱中储存数小时,已完成内源性多肽水解过程[23],为了减少检测结果的变异情况,建议采用晨尿的中段尿。尿液多肽在常温下保存≤6 h时组成和浓度相对稳定,≤-20℃可保存数年,期间避免反复冻融[24]。尽管饮食、锻炼和饮水等可影响尿液多肽组,但相对而言,尿液是一种较为稳定的样本[5]。同时尿液采样非侵入性,可重复采集,尿液中蛋白含量较低,且相对分子质量<10 000的多肽高度富集[25],这可能与小分子多肽更易通过肾小球滤过屏障有关[26],这种特性降低了后续多肽分离富集和检测的难度,也降低了高丰度蛋白对潜在蛋白/多肽指纹图谱检测信号的干扰程度[27]。因此尿液是多种疾病相关蛋白/多肽指纹图谱检测相对理想的样本之一,尤其是泌尿系统肿瘤。
脑脊液是充满在各脑室、蛛网膜下腔和脊髓中央管内的无色透明液体,维持颅内压稳定。脑脊液多肽主要来源于中枢神经系统分泌和血液中穿过血-脑脊液屏障的小分子[10-11],被认为是神经系统疾病标志物的重要来源。基于脑脊液蛋白/多肽指纹图谱可用于神经退行性疾病、创伤性脑损伤、精神性疾病和脑肿瘤等的检测[28-30]。然而脑脊液采样存在侵入性,不易获取,使得基于脑脊液蛋白/多肽指纹图谱的检测技术在临床应用中受到一定限制。
唾液是由分布在口腔内的主要唾液腺(腮腺、颌下腺和舌下腺)和其他腺体分泌的一种液体,采样具有非侵入性的优势,基于唾液的蛋白/多肽指纹图谱可进行口腔疾病的检测,如口腔癌[31]。然而由于蛋白进入唾液中后即发生降解,且在唾液采集后降解过程依旧存在,使得唾液多肽组检测结果的重复性大大降低。其他变量因素如年龄、性别和饮食等也会影响唾液多肽组[32]。这些因素加大了唾液蛋白/多肽指纹图谱临床应用的挑战。
其他体液样本,包括泪液和浆膜腔积液等,也有关于蛋白/多肽指纹图谱的研究报道。泪液样本收集具有非侵袭性的优势,泪液多肽组在同一个体不同时间相对稳定,然而个体间差异较大,增加了分析的复杂性[33]。重要的是泪液样本目前主要评估眼部疾病,如干眼症和睑板腺功能障碍等,在肿瘤中的研究相对较少[34-35]。浆膜腔积液,包括胸腔积液和腹腔积液等,与靶组织较近,关系密切,其中含有肿瘤细胞和微环境分泌的可溶性因子,通常与肿瘤晚期关系更加密切[36]。然而采样具有侵入性,早期检测的价值不足[37],重要的是肿瘤中浆膜腔积液蛋白/多肽指纹图谱研究相对较少,所报道的癌种较少,提供的证据和信息有限,因此仍需更多关于其蛋白/多肽指纹图谱的研究以支持其产业化发展和临床应用。
指导意见2:样本的选择与检测疾病类型和样本可及性有关。血液和尿液临床应用前景相对较好。各类样本采集后,应尽快分离检测组分,并低温保存,避免反复冻融,以增加结果的准确度和可重复性。
2.2 多肽的提取
检测组分中多肽组检测前,需对多肽进行有效提取。而多肽易被蛋白酶/肽酶降解,导致蛋白/多肽指纹图谱中疾病相关信息减弱甚至丧失,因此体外样本预处理时间应尽可能缩短[38]。在检测组分保存前和多肽提取前,加入特异性和(或)广谱性蛋白酶/肽酶抑制剂至样本中,可抑制蛋白酶/肽酶活性,降低多肽水解程度[39]。然而有些蛋白酶/肽酶抑制剂就是多肽,或者是多肽类似物,加入后丰度相对较高,不仅增加了样本中多肽的变异性,而且对内源性多肽分析起到抑制作用[2],因此需慎重考虑。除了加入蛋白酶/肽酶抑制剂外,收样后快速冷冻是另一种方法,目的也是降低蛋白酶/肽酶对多肽的降解,同时减少冻融次数,并在冰上进行多肽提取[18,31]。
另外生物样本中多肽丰度相对较低,例如血清中小分子多肽占比<1%,其他22种大分子蛋白如清蛋白、α-1抗胰蛋白酶、α-2巨球蛋白、转铁蛋白和γ-球蛋白等占比达99%以上[40],在质谱检测过程中易覆盖低丰度多肽信号。理想的多肽提取方法应尽可能捕获和富集更多多肽,尤其是疾病相关多肽,并去除高丰度干扰蛋白和其他小分子干扰物质。
目前一些方法已用于去除高丰度干扰蛋白和其他小分子干扰物质进而富集多肽,如超滤离心、选择性沉淀、固相萃取、磁珠法和免疫沉淀法等,对多肽的富集效率达到100~1000倍[41]。
超滤离心是最常用的多肽提取方法,运用超滤膜分离低相对分子质量多肽和高相对分子质量蛋白[42]。这种方法快速且易操作,然而可能导致直径大于滤膜孔径的多肽丢失,且难以去除直径小于滤膜孔径的其他小分子干扰物质,比如代谢分子和脂质,而这些小分子会降低质谱检测时多肽的电离效率,同时有些多肽易和滤膜结合,导致多肽的丢失[43]。
选择性沉淀法运用到多种样本中的多肽提取,具有快速、高效、经济和重复性好的特点[44]。选择性沉淀大分子高丰度蛋白,而低分子多肽和其他小分子干扰物质保留在溶液中。常用沉淀试剂包括有机溶剂(乙腈、丙酮和甲醇)、酸(三氟乙酸)或硫酸铵等[41],溶剂/酸和蛋白的作用很快,且能有效终止蛋白酶/肽酶活性。尽管沉淀法比较通用,但针对不同类型样本需选择合适的沉淀试剂,以提高多肽的提取效率[45],同时操作需小心,尽可能减少大分子蛋白聚集时捕获小分子多肽,导致检测结果的一致性下降[46]。另外沉淀法虽然能高效去除高丰度蛋白,但脂质和代谢分子仍在上清中存在,会干扰质谱检测时多肽的电离效率,有必要进一步处理。采用水不相溶的有机溶剂乙酸乙酯或乙醚等可有效地回收生物样本中多肽并去除其他小分子干扰物质[43,47],然而该方法重复性不足,目前只能作为实验室技术手段。
固相萃取利用选择性吸附与选择性洗脱的液相色谱法分离原理,可从复杂生物样本中提取多肽,并去除大分子蛋白干扰,以及不需要的小分子干扰物质和盐等[48],效果依赖于多肽和吸附剂的互作,也可用于沉淀法获得的样本中脂质和疏水性小分子物质的去除,提高多肽纯度[49]。这种方法显示出极佳的多肽提取效率和干扰物质清除效率,然而有些相具有过度选择性,不利于广谱的多肽组学研究,且很多时候发现仅对某个亚类多肽具有很好的富集效果。
磁珠法可用于复杂生物样本中的多肽富集和研究[50-51],分离富集多肽的效果理想,包括弱阳离子交换磁珠(weak cation exchange magnetic beads,MB-WCX, 参考http://www.purimagbead.com/Product/1894201745.html)、金属螯合磁珠(如铜离子螯合纳米磁珠, 参考http://www.purimagbead.com/Product/8910524421.html)和疏水磁珠(如疏水C8磁珠,参考http://www.purimagbead.com/Product/0548721933.html)等,其中MB-WCX在平均出峰量、平均峰面积和平均峰强度等方面均占优势。然而磁珠法多肽富集试剂价格较高,且以科研型试剂为主,大规模的临床应用前景受限。
免疫沉淀法利用特异性抗体与多肽结合,特异性高,能有效检测低丰度多肽[52-53],目前临床应用已有>50年的历史。该方法依赖于高特异性的抗体的开发,然而当前合适的高特异性的抗体有限[54],无法有效地对样本中的多肽进行全面分析。近年来,我国学者通过精确调控电化学蚀刻条件开发了具有纳米级孔道的多孔硅颗粒材料,能有效捕获富集小分子多肽而去除大分子高丰度蛋白,同时基于孔道排阻效应抑制蛋白酶/肽酶和小分子多肽的酶解接触,对多肽起到保护作用[55-56]。另外基于多肽不同的理化性质,研究者通过对多孔硅颗粒材料进行表面化学修饰,实现对血清中低丰度多肽广谱性和靶向性的捕获富集[57-58],且预处理步骤简单,有效地降低多肽的损失和临床应用时间消耗。基于该技术,建立了产业化的多孔硅颗粒材料生产体系和质量控制体系,开发了多款多肽提取试剂盒,并获得了国家药品监督管理局(National Medical Products Administration,NMPA)备案证,解决了多肽组学检测敏感度、稳定性和重复性问题,为蛋白/多肽指纹图谱相关检测产品研发、转化和临床应用奠定了基础。
指导意见3:生物样本中低丰度多肽高敏感度、高稳定性和高重复性的捕获富集是蛋白/多肽指纹图谱相关检测产品开发和临床应用的基础。基于多孔硅颗粒材料开发的多肽提取方法和试剂盒极具临床应用前景。
2.3 蛋白/多肽指纹图谱
检测技术基于多肽组学数据分析和筛选疾病相关多肽,能为特定疾病的诊断、治疗决策以及疗效和预后评估提供理想的方法[5]。多肽组学的检测常采用非靶向技术,以分析具有某特定疾病患者和不具有某特定疾病患者样本中所有多肽的差异[4,59]。与靶向检测技术比较,非靶向的多肽组学技术可同时分析众多多肽特征峰水平。这对于揭示复杂且异质性强的疾病极为关键。基于多个多肽特征峰的蛋白/多肽指纹图谱能显著增加检测的准确度[6-7]。质谱技术兼具高敏感度、高特异度和多指标联检的优势,目前已广泛用于多肽组学研究[60-61],是分析和发现疾病相关多肽的主要工具。该技术可根据不同多肽分子的质荷比(mass to charge ratio,m/z)微小差异高效检测复杂样本的蛋白/多肽指纹图谱[62-65]。
目前用于多肽组检测的质谱技术有多种,主要包括液相色谱串联质谱(liquid chromatography-tandem mass spectrometry,LC-MS/MS)、毛细管电泳质谱(capillary electrophoresis mass spectrometry,CE-MS)、表面增强激光解析电离飞行时间质谱(surface enhanced laser desorption ionization time of flight mass spectrometry,SELDI-TOF MS)和基质辅助激光解析离子化飞行时间质谱(matrix assisted laser desorption ionization time of flight mass spectrometry,MALDI-TOF MS)等[66]。
LC-MS/MS用于蛋白/多肽组检测,敏感度和准确度高,可检测上千种多肽,提供大量生物学信息[67-68],提升对复杂生物样本的分析能力。然而LC-MS/MS对检测组分前处理要求较高,有些脂质和代谢物相对分子质量和多肽类似,若没有被有效去除,会干扰LC-MS/MS对蛋白/多肽组的检测性能[48]。同时由于液相分离过程耗时长和通量低,临床应用时大批量样本检测受限。最关键的是LC-MS/MS自动化难度较大,操作繁琐,通常更适用于研发,无法将组学检测直接用于临床检验。截至2024年6月30日共有53款LC-MS/MS获得NMPA医疗器械注册证,基于LC-MS/MS技术获证的试剂盒也主要集中在维生素检测、治疗药物监测、激素类检测、儿茶酚胺类检测、胆汁酸检测和新生儿筛查等方面,均是基于定量方式检测单一或几个小分子应用于临床。
CE-MS常用于尿液蛋白/多肽组检测,可对低相对分子质量的多肽进行有效和高重复性的分析[69-70]。相对LC而言,CE的优势在于稳健性、可重复性和对高pH环境的抗干扰能力较高,增加了结果的可比性[71-72]。另外整个分析过程中缓冲液组成保持不变,这为待分析物电离提供了一致的环境[73-74]。然而CE-MS相对耗时,不适合大样本高通量检测,临床应用前景受到一定限制。
SELDI-TOF MS用于蛋白/多肽组检测已有许多报道,然而所使用的芯片有效表面积较小,对于小分子多肽富集不完全,不能完整地反映低丰度多肽所含有的生物学信息[75]。此外由于技术可重复性较差,易受多种因素影响,对于相同疾病的研究,不同实验室获得的结果较难达到一致,限制其临床应用的价值[76]。
MALDI-TOF MS是目前国产替代最成熟的质谱平台,是极具临床应用前景的质谱平台。与其他检测技术平台比较,MALDI-TOF MS用于蛋白/多肽组检测具有样本前处理要求低、样本量需求低、通量大、检测效率高、抗干扰能力强、自动化难度低和操作便捷等优势[77-79]。这使得MALDI-TOF MS可以全面识别生物样本中的多肽组学信息以发现疾病相关多肽特征峰。在过去十余年,基于该技术平台已发现多种肿瘤相关多肽[80-81]。MALDI-TOF MS成像可用于表征多肽组成以提供分子空间分布分析。该分析可与常规组织学数据相结合,分析感兴趣的组织学区域中数百个靶点的分布[82-83]。这为探索恶性疾病相关分子的空间分布异质性提供了最佳手段,比如多种肿瘤[84-85],可以区分不同亚型和不同级别肿瘤,从而为治疗决策提供信息。截至2024年6月30日共有44款MALDI-TOF MS获得NMPA注册证。基于MALDI-TOF MS技术获证的试剂盒应用领域包括微生物检测和基因检测等,其中微生物检测是基于质谱数据与数据库比对鉴定获得结果,这与基于蛋白/多肽指纹图谱的人工智能模型预测或者数据库比对来检测疾病的逻辑一致。在液体活检领域目前已有用于人体血清样本中蛋白/多肽指纹图谱采集的MALDI-TOF MS获得了NMPA注册证,可进行人体血清蛋白/多肽指纹图谱的检测。
指导意见4:质谱技术是蛋白/多肽指纹图谱检测的重要平台,其临床应用需重点考虑样本前处理要求、检测成本、检测效率、结果可重复性和操作便捷性等因素,其中MALDI-TOF MS在蛋白/多肽指纹图谱检测技术的转化和临床应用上具有一定的优势。
2.4 蛋白/多肽指纹图谱的检测数据分析
通常蛋白质组学中,质谱检测的MS/MS光谱采用生物信息学软件进行分析,包括PEAKS、ProteinPilot或MaxQuant等。这些软件利用不同的算法识别蛋白片段,然后在数据库中检索,识别所属的蛋白和鉴定序列。然而常规蛋白质组学研究通常采用特定的蛋白酶消化后进行质谱检测,比如采用胰蛋白酶,其N端或C端氨基酸位点比较一致。而多肽的N端和C端氨基酸序列受多种蛋白酶/肽酶消化的影响,比较复杂,基于传统的蛋白质组学数据库分析难以获得高准确度的结果。这对蛋白/多肽指纹图谱的检测数据分析带来了极大挑战。一种方法是检索SATPdb和SwePep等多肽组学数据库进行数据分析。这些数据库包含了最新研究发现的治疗性或内源性多肽[86-87]。另外多肽的翻译后修饰(post-translational modifications,PTMs)对于多肽和受体的互作和拮抗蛋白酶/肽酶的消化至关重要,常见的PTMs包括C端的酰胺化和N端的乙酰化,另外还有甲硫氨酸氧化以及精氨酸和谷氨酰胺脱酰胺。这些修饰给蛋白/多肽指纹图谱的检测数据分析带来了新的挑战。PERKS软件可对多肽及其PTMs进行有效分析[49]。在蛋白/多肽指纹图谱的检测数据分析过程中,需要重要考虑的一个因素是过滤搜索结果,此时应用到错误发现率阈值,因为实验发现的和理论光谱数都达到数千个,考虑到各种切割模式和PTMs修饰,错误匹配的可能性较大。为了降低这种可能性,通常采用和多肽氨基酸组成一致但序列翻转或打乱的诱饵进行检索,评估在数据库中检索时假阳性结果出现的可能分值[88]。
生物体内部分多肽具有生物活性,如细胞因子、生长因子和激素等。PeptideRanker和PeptideLocator可用于分析多肽序列是否具有生物活性[89-90]。比如PeptideRanker分值>0.5提示具有潜在生物活性。一些常见的多肽激素类物质在此软件中预测分值较高,然而仍有部分假阴性和假阳性结果,需慎重对待。
多肽组学检测为疾病相关蛋白/多肽指纹图谱的构建提供了基础。许多单一多肽用于疾病检测的准确度有限,尤其是面对分子机制和特征复杂、异质性极强的疾病时,限制了其应用。通过人工智能整合多个多肽特征峰的蛋白/多肽指纹图谱可增加结果的稳定性、准确度和降低变异系数[91-93],同时特异度也显著提高[92]。蛋白/多肽指纹图谱模型需依据《人工智能医疗器械注册审查指导原则》构建[94]。
首先应建立明确的训练集、验证集和测试集队列,每个队列中总样本量和各分组样本量的计算可参考原国家食品药品监督管理总局发布的《医疗器械临床试验设计指导原则》给出的方法进行计算[95],从而满足抽样误差的要求,在条件允许的情况下尽量提高样本量。队列内分组比例可参考全国流行病学统计数据,对于各分组比例未获得全国流行病学统计数据的疾病,队列设计可依据各机构掌握的先验数据设计分组的比例,队列内和队列间无样本重复;
其次由于不同地区和不同临床机构在人群组成、流行病学特征和样本采集操作等方面可能存在差异,为保证队列样本的代表性,各队列样本应来源不同省市有代表性的临床中心,以提高算法模型的泛化能力,降低偏倚;
再者,建议对各队列中各样本采集、预处理、标注和存储等环节建立规范流程和详细记录,形成体系化管理,保障样本的稳定性。对于体外诊断产品的开发,需根据临床信息,如病理结果、镜检结果、影像结果和其他标志物结果等,来标注阴阳性分类和亚型分类等。同时,可根据产品预期使用人群、地区和流行病学特征,对其他生理异常或病变进行标注,作为算法训练的标签。样本收集和临床信息获取应获得伦理批准或者豁免。样本的采集和保存,以及信息的传输和使用应符合《中华人民共和国网络安全法》、《科技部人类遗传资源管理办法》和原国家食品药品监督管理总局《医疗器械网络安全注册技术审查指导原则》等法律法规的要求[96-98];
随后,收到的样本应建立质量控制标准,剔除不合格样本。多肽组的检测需制定标准操作程序(standard operating procedure,SOP),按照SOP获取各样本多肽组学数据,并针对多肽组学数据建立质量控制标准,剔除不合格数据,筛选满足质量要求的数据,特别是对于质控样本的多肽检测信号的变异控制,建议批间变异系数≤15%;
最后由于多肽和疾病的关系复杂,疾病精准检测的蛋白/多肽指纹图谱构建离不开数据的标准化处理和人工智能技术的应用[99-100]。多肽组学数据预处理及归一化后,采用机器学习的方法建立包含多个多肽特征峰的集成模型。机器学习算法包括逻辑回归、随机森林(random forest,RF)和支持向量机(support vector machine,SVM)等[101-103]。基于标准化数据处理和机器学习算法建立的模型,需通过测试集验证后完成模型定型,并建立判别诊断软件。包含定型模型的诊断软件需进一步通过建模后新采集的外部独立测试队列数据进行模型性能测试,并获取模型性能评估结果。诊断软件应用前需进一步在与预期临床应用场景相吻合的人群队列中开展前瞻性临床研究(图1)。
指导意见5:疾病相关蛋白/多肽指纹图谱模型构建需依据《人工智能医疗器械注册审查指导原则》[94],基于多家代表性临床中心大样本建立符合统计学和流行病学的训练集、验证集和测试集。样本采集和信息收集应在符合法律法规的基础上建立体系化管理体系。样本和多肽组学数据需建立质量控制标准明确是否符合要求。多肽组的检测应采用统一的操作标准进行,对多肽组学数据应进行标准化处理并结合人工智能算法进行疾病相关多肽筛选和精准检测模型构建。模型性能需通过独立测试队列和前瞻性队列进行评估。
3. 蛋白/多肽指纹图谱检测的肿瘤临床应用
3.1 蛋白/多肽指纹图谱检测用于肿瘤诊断
肿瘤诊断包括早筛和辅助诊断。目前已有基于多肽的产品在肿瘤诊断中的应用,如用于小细胞肺癌检测的胃泌素释放肽前体(pro-gastrin releasing peptide,Pro-GRP)[104]和用于甲状腺髓样癌检测的降钙素等[105]。2009年获得美国食品药品管理局(Food and Drug Administration,FDA)批准用于评估有盆腔包块的患者罹患卵巢癌风险的OVA1TM测试,包括糖类抗原125(carbohydrate antigen 125,CA125)、甲状腺素运载蛋白、载脂蛋白A1、β2-微球蛋白和转铁蛋白,其中甲状腺素运载蛋白和载脂蛋白A1是多肽分子,特异性高,是主要定性标志物[106-108]。这些应用初步显示了多肽在临床肿瘤诊断中的价值[109]。目前在多种肿瘤中,研究者广泛开展了蛋白/多肽指纹图谱检测产品的研发和验证工作(表3)[56,110-114]。
肺癌是我国发病率和死亡率均位居第1位的恶性肿瘤[115]。早期筛查和精准检测有助于降低肺癌发病率、死亡率和医疗负担[116-117]。通过MALDI-TOF MS检测非小细胞肺癌血清多肽组,发现11个差异多肽,基于遗传算法(genetic algorithm,GA)建立的模型包含2个多肽特征峰,敏感度和特异度分别高达92.9%和91.7%[118]。另外也有学者通过MALDI-TOF MS检测血清多肽组,基于K-近邻算法(K-nearest neighbor,KNN)建立包含5个差异性多肽的非小细胞肺癌诊断模型,测试集敏感度和特异度分别高达80.7%和91.2%[119]。近期,研究者基于ClinMS-PlatⅠ血清多肽指纹图谱检测技术,纳入了12家大型临床中心的11 345例肺癌队列样本进行肺癌的血清蛋白/多肽指纹图谱检测,对于肺部结节患者同时结合CT影像特征数据,进行人工智能分析,建立模型。模型经测试集评估敏感度和特异度分别高达90.4%和97.6%,其中原位癌检出率85.7%,Ⅰ期肺癌检出率高达90.8%,针对临床难判结节相对集中的5~15 mm肺结节的检测准确度高达93.2%,展现出优异的性能[56]。目前该项研究成果已实现定型和产品化,可以优化肺癌的低剂量螺旋CT检测中的假阳性和漏检问题[120],为肺癌和肺结节的临床检测提供诊疗参考。
图1 蛋白/多肽指纹图谱检测技术建立流程图
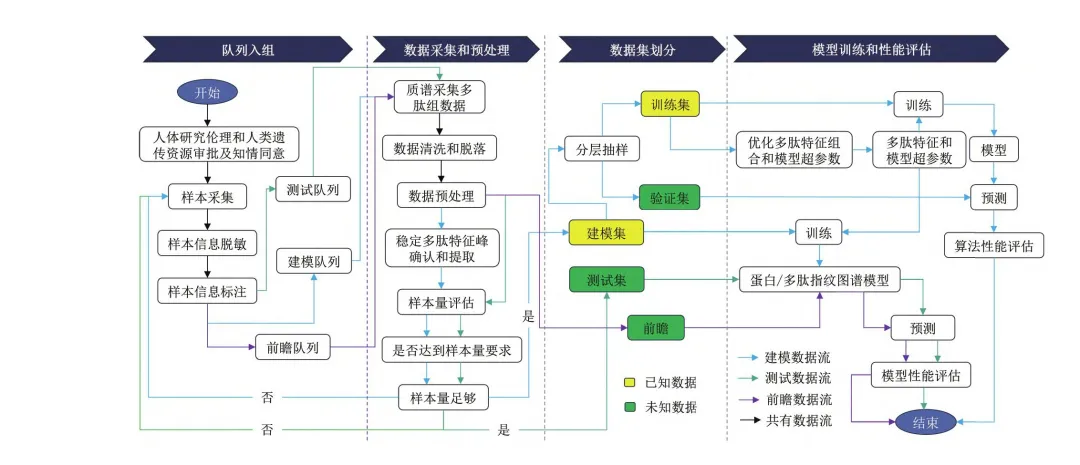
表3 蛋自/多肽指纹图谱在肿瘤诊断中的主要研究及其阶段

注MALDI-TOF MS:基质辅助激光解析离子化飞行时间质谱;LDT:实验室自建项目;CE-MS:毛细管电泳质谱;NA:缺失值(not available)
消化系统肿瘤在我国发病率较高,其中结直肠癌、肝癌、胃癌、食管癌和胰腺癌五大癌种均位居前十,且是除肺癌外死亡率最高的五大癌种[115],早筛早诊对于消化系统五癌防控意义重大[121-125]。目前指南和专家共识推荐的消化系统五癌筛查和早诊技术以内镜学和影像学为主[122-124,126-127],存在侵入性或辐射危害,依从性低,资源有限,临床急需新的液体活检技术对消化系统五癌高危人群进行富集,减少不必要的内镜学或影像学检查,提高高危人群依从性,降低医疗负担。有学者基于多肽组学数据和人工神经网络(artificial neural network,ANN)构建了包含4个多肽特征峰的结直肠癌检测模型,敏感度和特异度分别高达91%和93%[128]。另一个基于5个多肽特征峰建立的模型诊断结直肠癌的敏感度和特异度分别高达82%和93%[129]。基于MALDI-TOF MS平台检测多肽组,采用逻辑回归建立的包含5个多肽的诊断模型诊断结直肠癌敏感度和特异度分别高达95.6%和87.9%[130]。基于尿液多肽组学数据建立的包含31个多肽特征峰的模型对肝细胞癌和非肝细胞癌人群(包括肝硬化、非肝硬化肝疾病和健康人群)判别的敏感度和特异度分别为79.5%和85.1%[110]。基于多肽组学数据和径向基函数神经网络(radial basis function neural network,RBFNN)建立的包含6个多肽特征峰的模型用于识别肝细胞癌骨转移和未转移患者的敏感度和特异度分别为85.3%和85.7%,受试者工作特征曲线(receiver operating characteristic,ROC)曲线下面积为0.911[81]。基于多肽组学数据,通过GA、有监督神经网络(supervised neural network,SNN)和快速分类(quick classifier,QC)算法构建的蛋白/多肽指纹图谱诊断模型,诊断胃癌的平均敏感度和特异度分别为79.3%和86.5%[111]。采用QC算法构建的包含5个多肽特征峰的食管鳞状细胞癌诊断模型的敏感度和特异度分别高达98.5%和97.2%[112]。采用经多肽组学数据分析发现的m/z为3 884和5 959的2个多肽建立的模型用于识别胰腺癌,敏感度和特异度分别高达86.3%和97.6%。在常规肿瘤标志物CA19-9和癌胚抗原(carcinoembryonic antigen,CEA)检测过程中,加入m/z为3 884的多肽,可有效提升结果的准确度[131]。这些研究表明,蛋白/多肽指纹图谱在消化系统五癌筛查和辅助诊断中的巨大潜力,然而尚需开展多中心大样本的研究验证其性能。
近期,研究者基于ClinMS-PlatⅠ血清多肽指纹图谱检测技术,纳入12家不同中心的11 097例结直肠癌队列临床样本进行结直肠癌的血清蛋白/多肽指纹图谱的建模、测试和验证。经验证,结直肠癌血清多肽指纹图谱模型的敏感度和特异度分别高达85.8%和89.6%,其中早期结直肠癌检出率高达85.6%,显示早诊的特性,有助于降低结直肠癌发病率、死亡率和医疗负担[56]。目前该项研究成果已实现定型和产品化。另外在泌尿系统肿瘤中,基于尿液多肽组学数据和KNN算法筛选的5个多肽特征峰建立的模型对膀胱癌诊断的敏感度高达93.4%,其中早期检出率90.0%,在健康人群和血尿症患者中的特异度分别为97.8%和73.3%[132]。有研究分析721例膀胱癌患者尿液多肽组,基于SVM建立了包含116个多肽的蛋白/多肽指纹图谱模型,用于膀胱癌检测,经大样本验证后显示,对于膀胱癌的敏感度和特异度分别为91.1%和67.6%[113]。在生殖系统肿瘤中,基于多肽组学数据来源的蛋白/多肽指纹图谱可有效区分Ⅰ期卵巢癌患者和非卵巢癌人群[133]。序列为VSFELFADK和FEDENFILK的2个多肽在卵巢癌患者血浆中水平增加,检测卵巢癌的特异度均为100%,敏感度分别为75%和78.6%[134]。基于血清中3个差异多肽[谷氨酸-酪氨酸(Glu-Trp)、羟脯氨酸-亮氨酸(Hyp-Leu)和苯丙氨酸-苯丙氨酸(Phe-Phe)]检测早期上皮性卵巢癌的敏感度和特异度分别为63.6%和84.5%,虽然与肿瘤标志物CA125(敏感度81.8%,特异度63.8%)性能相似,但与CA125联合后敏感度和特异度分别高达80.4%和94.4%[135]。在前列腺癌中,分析823例前列腺特异性抗原(prostate specific antigen,PSA)<15 ng/mL患者的尿液多肽组,基于SVM建立了包含19个差异多肽的蛋白/多肽指纹图谱模型。在280例前列腺癌外部验证队列中发现,该模型对高级别前列腺癌以及低级别前列腺癌和(或)非泌尿系统疾病的敏感度和特异度分别为89.6%和59.1%[114]。在乳腺癌中,基于SVM筛选的3个差异性多肽特征峰建立的诊断模型的敏感度和特异度分别高达91.9%和91.7%,而传统乳腺癌标志物CA15-3对同一人群的敏感度和特异度仅为41.7%和43.2%[136]。
除了在单癌种筛查和辅助诊断中的研究外,在多癌种检测中,蛋白/多肽指纹图谱检测也显示出优异性能。基于MALDI-TOF MS检测和无监督聚类分析的研究发现,血清中651个多肽可有效区分前列腺癌、乳腺癌、膀胱癌和对照人群,并且在3种肿瘤中血清蛋白/多肽指纹图谱特征不同[137]。近期,研究者基于ClinMS-PlatⅠ血清多肽指纹图谱检测技术,纳入14家临床中心11 921例样本,建立了包括结直肠癌、肝癌、胃癌、食管癌和胰腺癌消化系统五大癌种的检测模型,模型的敏感度和特异度分别高达82.3%和89.7%,早期检出率也高达81.3%,同时癌种溯源综合准确度达到83.1%[56]。
指导意见6:蛋白/多肽指纹图谱检测为肿瘤检测提供了一种高效、便捷、精准且具有广泛应用潜力的方法。初步实现了对肺部肿瘤和结直肠肿瘤的早期检测、肺结节的良恶性鉴别和消化系统五大癌种(结直肠癌、肝癌、胃癌、食管癌和胰腺癌)的联合检测。其他基于蛋白/多肽指纹图谱检测的产品处于基础研究层面,尚未成熟。
3.2 蛋白/多肽指纹图谱检测用于肿瘤精准治疗决策
指导蛋白/多肽指纹图谱检测同样可用于肿瘤精准治疗决策指导(表4)[138-146]。2007年,Taguchi等[138]使用MALDI-TOF MS检测非小细胞肺癌患者的血清样本获得多肽组学数据,基于其中8个差异多肽建立模型,可有效预测非小细胞肺癌患者使用表皮生长因子受体酪氨酸激酶抑制剂(epithelial growth factor receptor tyrosine-kinase inhibitors,EGFR-TKIs)后的临床预后情况。随后这个模型被应用于商品化检测项目VeriStrat。该项目的效果已在非小细胞肺癌的多项个体化治疗研究中得到验证[139-140],并通过FDA审核,被纳入2016年的美国国立综合癌症网络指南建议[141-143]。一个基于血浆多肽组学数据和RF算法建立的包含3个多肽的模型有效区分局部进展期直肠癌患者术前新辅助放化疗的敏感性,敏感度达到77.8%,特异度为91.7%,总体准确度为85.7%[144]。我国研究者通过分析晚期肺鳞癌患者接受紫杉醇联合铂类化疗前血液样本多肽组,基于5个差异多肽特征峰建立了晚期肺鳞癌患者接受紫杉醇联合铂类化疗方案的敏感性预测模型,敏感度为90.0%,特异度为80.0%[145]。转移性黑色素瘤预后较差,免疫治疗作为一线或二线可选的治疗方案能有效改善患者无进展生存期和总生存期,然而仅30%的患者对免疫治疗有反应,传统采用免疫组织化学检测程序性死亡配体1(programmed cell death-ligand 1,PD-L1)难以有效筛选出对免疫治疗响应人群。有研究采用MALDI-TOF MS分析免疫治疗前转移性黑色素瘤患者血清样本多肽组发现,基于其中209个多肽特征峰的蛋白/多肽指纹图谱能有效预测患者对免疫治疗的敏感性,并经过5组共170例转移性黑色素瘤患者验证,HR=0.15(95%CI:0.06~0.40)[146]。
表4 蛋白/多肽指纹图谱在肿瘤精准治疗决策指导中的主要研究及其阶段

注EGFR-TKIs:表皮生长因子受体酪氨酸激酶抑制剂;MALDI-TOF MS:基质辅助激光解析离子化飞行时间质谱;NCCN:美国国立综合癌症网络;
指导意见7:蛋白/多肽指纹图谱检测已成功应用于评估EGFR野生型非小细胞肺癌患者使用EGFR-TKIs的预后情况,辅助临床做出治疗决策。一些蛋白/多肽指纹图谱检测与直肠癌和黑色素瘤等的治疗敏感性有关,但仍需更多临床试验进一步验证。
3.3 蛋白/多肽指纹图谱检测用于肿瘤患者疗效和预后评估
肿瘤治疗后,需对其疗效和预后进行监测,评估治疗效果和及时发现复发情况。有研究通过硅纳米材料试剂盒捕获多肽结合MALDI-TOF MS建立模型检测蛋白/多肽指纹图谱可以预测转移性结直肠患者对标准一线化疗方案的治疗反应,并发现间-alpha-胰蛋白酶抑制剂重链H4(interalpha-trypsin inhibitor heavy chain 4,ITIH4)及其肽段与结直肠癌对5-FU的耐药性相关[147]。另有研究发现,多肽Pro-GRP不仅可用于小细胞肺癌的诊断,同样可在其复发监测中发挥作用,敏感度和特异度分别为70%和91%[148]。有学者分析636例膀胱癌患者尿液多肽组,基于SVM建立了包含106个多肽的蛋白/多肽指纹图谱模型。该模型对膀胱癌复发预测的敏感度和特异度分别高达87%和57%[113];随后经有随访结果的331例患者验证,显示出良好的预后评估价值(HR=3.15,95%CI:1.73~5.70)[149]。采用MALDI-TOF MS检测揭示乳腺癌血清多肽组的研究发现,纤维蛋白原α链(fibrinogen alpha chain,FGA) 605-629、ITIH 347-356和载脂蛋白A2(apolipoprotein A2,APOA2) 43-52共3个多肽组合模型可用于乳腺癌的诊断和评估预后情况[150]。
指导意见8:蛋白/多肽指纹图谱检测在肿瘤患者疗效和预后评估中的应用潜力有待挖掘,目前处于基础研究阶段,仍需大量的临床数据支持,以扩展至更多癌种。
4. 小结与展望
肿瘤是全球人类死亡的主要原因之一,对其进行精准检测是提高疗效和降低发病率、死亡率与医疗负担的有效方式。目前,蛋白/多肽指纹图谱研究正处于蓬勃发展阶段,蛋白/多肽指纹图谱的检测分析方法和临床应用正日益受到重视,并在医学领域展现出巨大的潜力,多款多肽检测产品的临床应用显示了良好的价值。我国研究者近年来也基于蛋白/多肽指纹图谱推出了多款肿瘤检测产品,包括单癌种和多癌种。
未来期望更多的研究关注以下方面以拓展蛋白/多肽指纹图谱在临床肿瘤防控中的应用:
(1)深入揭示肿瘤中多肽的表达机制,研究不同多肽在肿瘤的发生、发展以及对治疗的敏感性与耐药性的影响和机制,为后续蛋白/多肽指纹图谱检测产品的开发提供更加扎实的理论依据;
(2)技术层面,开发新型多肽捕获富集和保护技术和检测技术,提升检测结果的稳定性、可重复性和准确度,建立符合临床要求的检测流程,为临床应用奠定基础;
(3)利用机器学习和深度学习算法对多肽组学数据进行分析,从海量的多肽组学数据中识别出具有诊断或预后意义的多肽特征峰,将多种多肽特征峰组合建立蛋白/多肽指纹图谱模型,以及与其他生物标志物结合,或组学数据整合,构建更可靠的模型;
(4)开展多中心大样本的临床研究,综合评估模型性能,并在预期应用场景中开展前瞻性研究,以及真实世界评估对人群肿瘤发病率与死亡率的影响和经济效益情况。本专家共识全面梳理了蛋白/多肽指纹图谱的检测分析技术、临床应用及其在肿瘤管理中的潜力,提出了一系列具有前瞻性和实践指导意义的共识推荐。未来,期待本专家共识进一步落地和实践,助力蛋白/多肽指纹图谱检测产品的研发、转化和临床应用,为居民的健康保驾护航。
专家组成员(按姓名汉语拼音字母排序):陈湖光(浙江大学医学院附属第一医院)、陈益定(浙江大学医学院附属第二医院)、丁小文(浙江省肿瘤医院)、范钰(江苏大学附属人民医院)、高越(杭州市第一人民医院)、韩明勇(深圳大学附属华南医院)、何正富(浙江大学医学院附属邵逸夫医院)、胡集祎(上海市质子重离子医院)、黄凌(广东省人民医院)、姜武(中山大学肿瘤防治中心)、梁文华(广州医科大学附属第一医院)、梁霄(浙江大学医学院附属邵逸夫医院)、刘建(浙江省中医药大学附属第二医院)、沈虹(浙江大学医学院附属第二医院)、宋章法(浙江大学医学院附属邵逸夫医院)、谈洁(湖南大学化学化工学院)、唐勇(广西医科大学附属肿瘤医院)、王彩琴(湖南省肿瘤医院)、邬建敏(浙江大学化学系)、熊斌(武汉大学中南医院)、余捷凯(浙江大学医学院附属第二医院)、袁瑛(浙江大学医学院附属第二医院)、翟晓慧(中山大学附属第六医院)、张大(郑州大学第一附属医院)、张燕(中山大学附属第六医院)
参考文献
[1] Li L, Wu J, Lyon CJ, et al. Clinical peptidomics:advances in instrumentation, analyses, and applications[J]. BME Front, 2023, 4:0019.
[2] Maes E, Oeyen E, Boonen K, et al. The challenges of peptidomics in complementing proteomics in a clinical context[J]. Mass Spectrom Rev, 2019, 38(3):253-264.
[3] Cabrera-Quio LE, Herberg S, Pauli A. Decoding sORF translation-from small proteins to gene regulation[J].RNA Biol, 2016, 13(11):1051-1059.
[4] Figueiredo D, Cruz RGB, Normando AGC, et al. Peptidomics strategies to evaluate cancer diagnosis, prognosis,and treatment[J]. Methods Mol Biol, 2024, 2758:401-423.
[5] Latosinska A, Frantzi M, Siwy J. Peptides as"better biomarkers"? Value, challenges, and potential solutions to facilitate implementation[J]. Mass Spectrom Rev, 2024,43(6):1195-1236.
[6] Mavrogeorgis E, Mischak H, Latosinska A, et al. Reproducibility evaluation of urinary peptide detection using CE-MS[J]. Molecules, 2021, 26(23):7260.
[7] Dakna M, Harris K, Kalousis A, et al. Addressing the challenge of defining valid proteomic biomarkers and classifiers[J]. BMC Bioinformatics, 2010, 11:594.
[8] Anderson NL, Anderson NG. The human plasma proteome:history, character, and diagnostic prospects[J]. Mol Cell Proteomics, 2002, 1(11):845-867.
[9] Thomas S, Hao L, Ricke WA, et al. Biomarker discovery in mass spectrometry-based urinary proteomics[J]. Proteomics Clin Appl, 2016, 10(4):358-370.
[10] Begcevic I, Brinc D, Drabovich AP, et al. Identification of brain-enriched proteins in the cerebrospinal fluid proteome by LC-MS/MS profiling and mining of the Human Protein Atlas[J]. Clin Proteomics, 2016, 13:11.
[11] Vitorino R. Digging deep into peptidomics applied to body fluids[J]. Proteomics, 2018, 18(2):1700401.
[12] Decramer S, Gonzalez de Peredo A, Breuil B, et al. Urine in clinical proteomics[J]. Mol Cell Proteomics, 2008,7(10):1850-1862.
[13] Magalhães P, Pontillo C, Pejchinovski M, et al. Comparison of urine and plasma peptidome indicates selectivity in renal pep
- 上一篇:蛋白质组学基础知识 2025/1/11
- 下一篇:不同检测中心的纳米粒子表面蛋白冠检测差异性分析——重复性分析 2025/1/6

